
Figure 1.
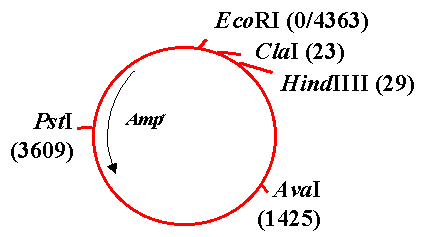
Figure 2. Le plasmide pBR322 est linéaire et constitué de 4363 paires de bases.
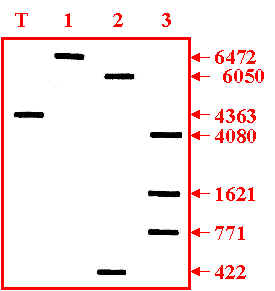
Figure 3.
TD 5 et 6 : GENETIQUE MOLECULAIRE -
EXERCICE 1.
1. Représenter les extrémités des fragments de restriction obtenus après digestion par les enzymes de restrictions suivantes en les classant selon la nature de ces extrémités (franches, 5'- ou 3'-sortantes).
BamH1 G/GATCC
HincII GTPy/PuAC
HpaII C/CGG
MspI C/CGG ou Cm/CGG (m :méthylée).
MboI /GATC
Sau3A GATC/
SmaI CCC/GGG
AccI GT/CGAC
KpnI GGTAC/C
EcoRI G/AATTC
PstI CTGCA/G
BglII A/GATCT
EXERCICE 2.
1. Si on utilise une enzyme de restriction possédant un site de restriction de 4 bases, quelles seront la fréquence de coupure d'un ADN génomique et la taille moyenne des fragments de restriction obtenus ?
2. Même question pour une enzyme ayant un site de reconnaissance de 6 paires de bases.
QUESTION 3.
On veut digérer par l'enzyme EcoRI, 10 µg d'un fragment d'ADN linéaire aux extrémités SmaI-KpnI. L'ADN est disponible en solution à la concentration de 0,5µg/ml. L'enzyme est à votre disposition à la concentration de 5U/µl dans une solution de glycérol 50%.
Sachant que :
- une unité (U) d'enzyme digère 1µg d'ADN en 1h à 37°C. Pour être certain que la coupure sera totale on utilise l'enzyme EcoRI 5 fois en excès.
- Pour que l'enzyme ait son maximum d'efficacité, il est nécessaire d'ajouter un tampon de digestion contenant du NaCl à 50mM, du MgCl2 à 10mM dans un tampon Tris 0,1M, pH 7,5. Vous disposer d'une solution de tampon de digestion concentrée (NaCl 0,5M; 0.1M; Tris 1M, pH 7,5).
- Pour que l'enzyme fonctionne correctement, la concentration en glycérol ne doit pas excéder 5%.
1. Décrire le protocole de digestion en respectant les conditions.
Après une incubation de 2 heures à 37°C, les produits de digestion sont analysés par électrophorèse sur gel d'agarose (figure 1).
2. Expliquer le principe de cette expérience.
3. Comment visualise t-on les fragments d'ADN sur le gel ?
4. Calculer la taille des différents fragments d'ADN produits de la digestion (puits 1) en utilisant les fragments d'ADN marqueurs de taille (puits 2). Ces fragments proviennent de la digestion par HindIII de l'ADN du phage Lambda [sept bandes sont visibles de tailles 23 - 9,5 - 6,6 - 4,3 - 2,3 - 2 et 0,56 kb (1 kb = 1000 paires de bases)].
La même expérience est réalisée avec d'autres enzymes et à donner les résultats suivants
Enzyme(s) utilisée(s) |
Taille des fragments obtenus (en kb) |
MspI |
8 et 0,5 |
EcoRI + MspI |
3,5 - 3 - 1,5 - 0,5 |
BglII |
7,5 et 1 |
EcoRI + BglII |
4 - 3 - 1 et 0,5 |
5. Donner la taille et la carte de restriction de ce fragment d'ADN sachant que le site MspI et situé près du site SmaI.
On veut insérer dans le vecteur plasmidique pUC les fragments EcoRI-EcoRI (fragment A), SmaI-MspI (fragment B) et BglII-KpnI (Fragment C) produits de la digestion de l'ADN initial..
L'ADN du vecteur pUC possède un SMC (site multiple de clonage). Les sites de restriction du SMC sont dans l'ordre suivant : EcoRI - KpnI - SmaI - BamHI - AccI - PstI - HindIII. Ces sites sont uniques dans le vecteur. On sait par ailleurs que le vecteur pUC possède 13 sites de reconnaissance par l'enzyme MspI et aucun pour l'enzyme BglII.
6. Quelles enzymes peut-on utiliser pour insérer les différents fragments A, B et C dans le SMC du vecteur pUC ?
Après insertion des fragments dans le plasmide, la construction plasmidique est introduite dans des bactéries (transformation) et un clone de bactéries transformées et mis en culture. L'ADN est ensuite purifié et on cherche à isoler les fragments clonés.
7. Quelles enzymes peut-on utiliser pour ressortir les fragments insérés dans le vecteur pUC ?
EXERCICE 4
Un fragment d'ADN est inséré au niveau d'un site HindIII du plasmide pBR322. La carte de restriction de ce plasmide non recombinant est présentée sur la figure 2.
Les produits de digestion enzymatique du plasmide recombinant sont analysés après électrophorèse sur gel d'agarose (figure 3). L'ADN du plasmide recombinant à été coupé par les enzymes EcoRI (puits 1), ClaI (puits 2) et PstI (puits 3). Le puits T montre les produits de digestion du plasmide pBR322 non recombinant digéré par l'enzyme EcoRI.
1. De combien de paires de bases est constitué le fragment d'ADN inséré dans le vecteur pBR322 ?
2. Positionner les sites de restriction ClaI et PstI de ce fragment sur un schéma.
Le plasmide recombinant est digéré par les enzymes de restriction ClaI et PstI.
3. Quelle sera la taille (en pb) des fragments produits de cette digestion ? Combien de bandes distinctes seront visualisées sur un gel d'agarose après une électrophorèse ?
4. Pourquoi n'a t-on pas inséré le fragment d'ADN au niveau du site AvaI de pBR322 ?
Figure 1. |
Figure 2. Le plasmide pBR322 est linéaire et constitué de 4363 paires de bases.
Figure 3. |